PYMOL241109変異蛋白解析
点変異の導入
中央に見えるトリプトファンをアスパラギン酸に変更します。
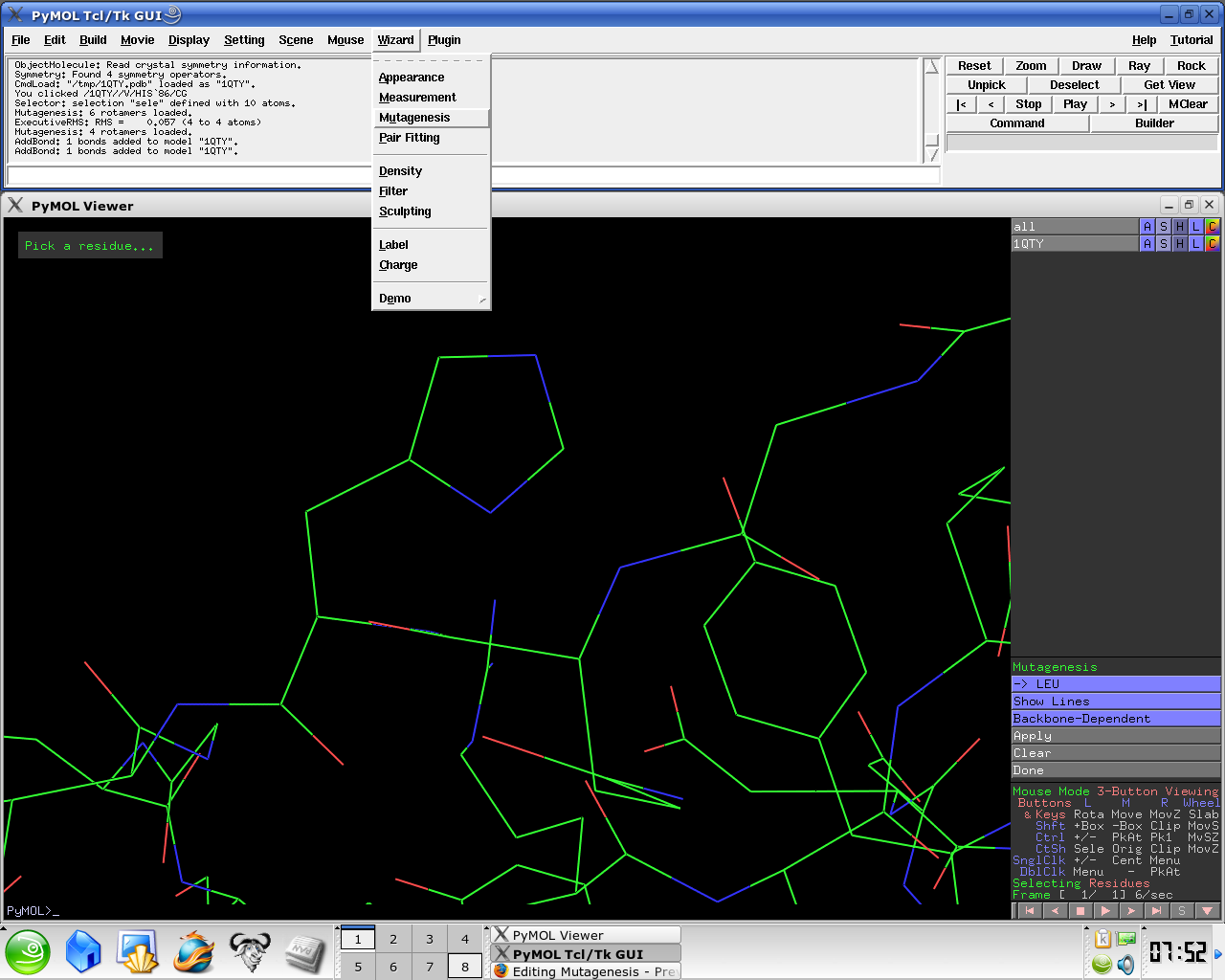
- Wizard → Mutagenesis

- 変異を入れたい残基をクリック
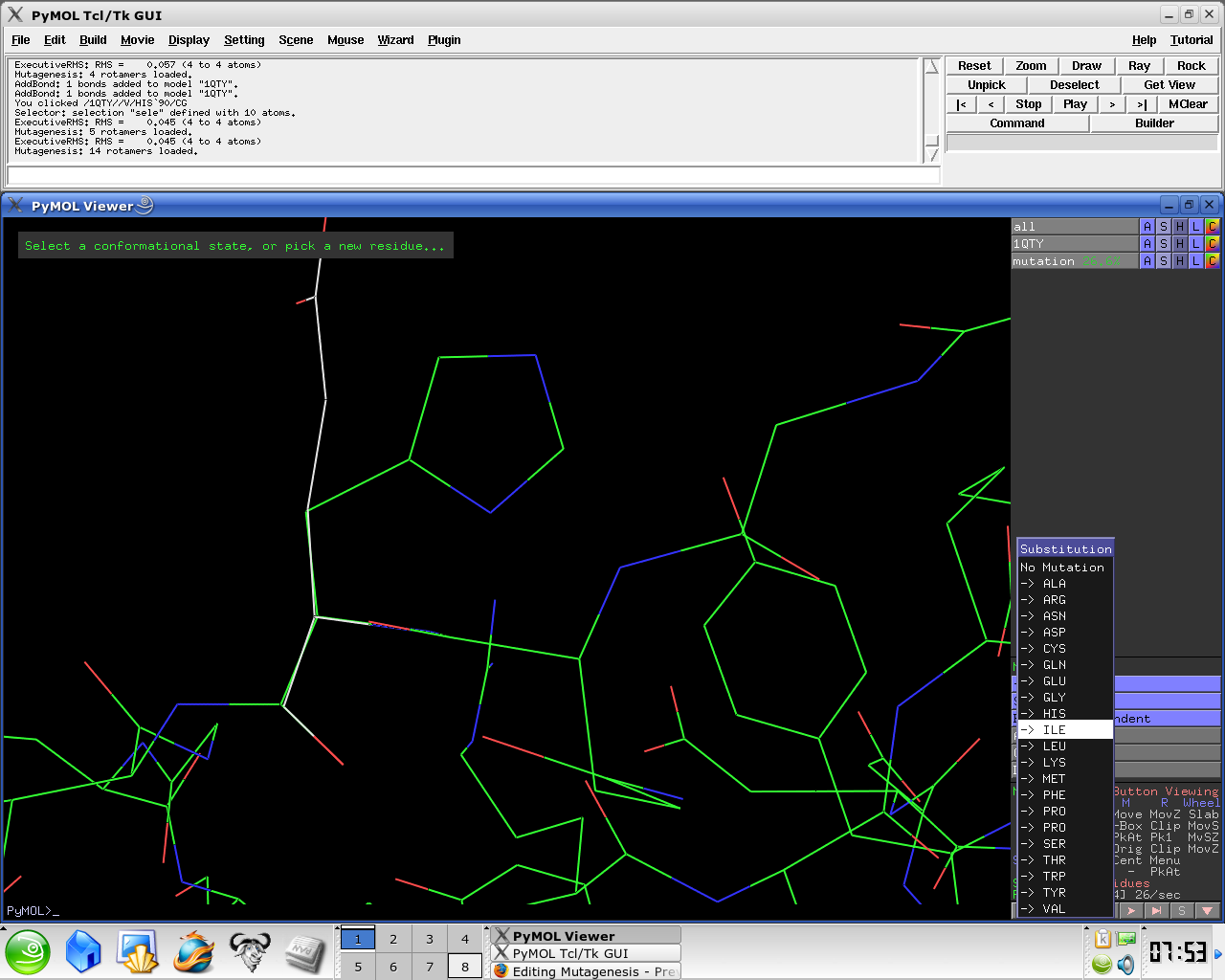
- "No mutation" から変更したい残基を選択

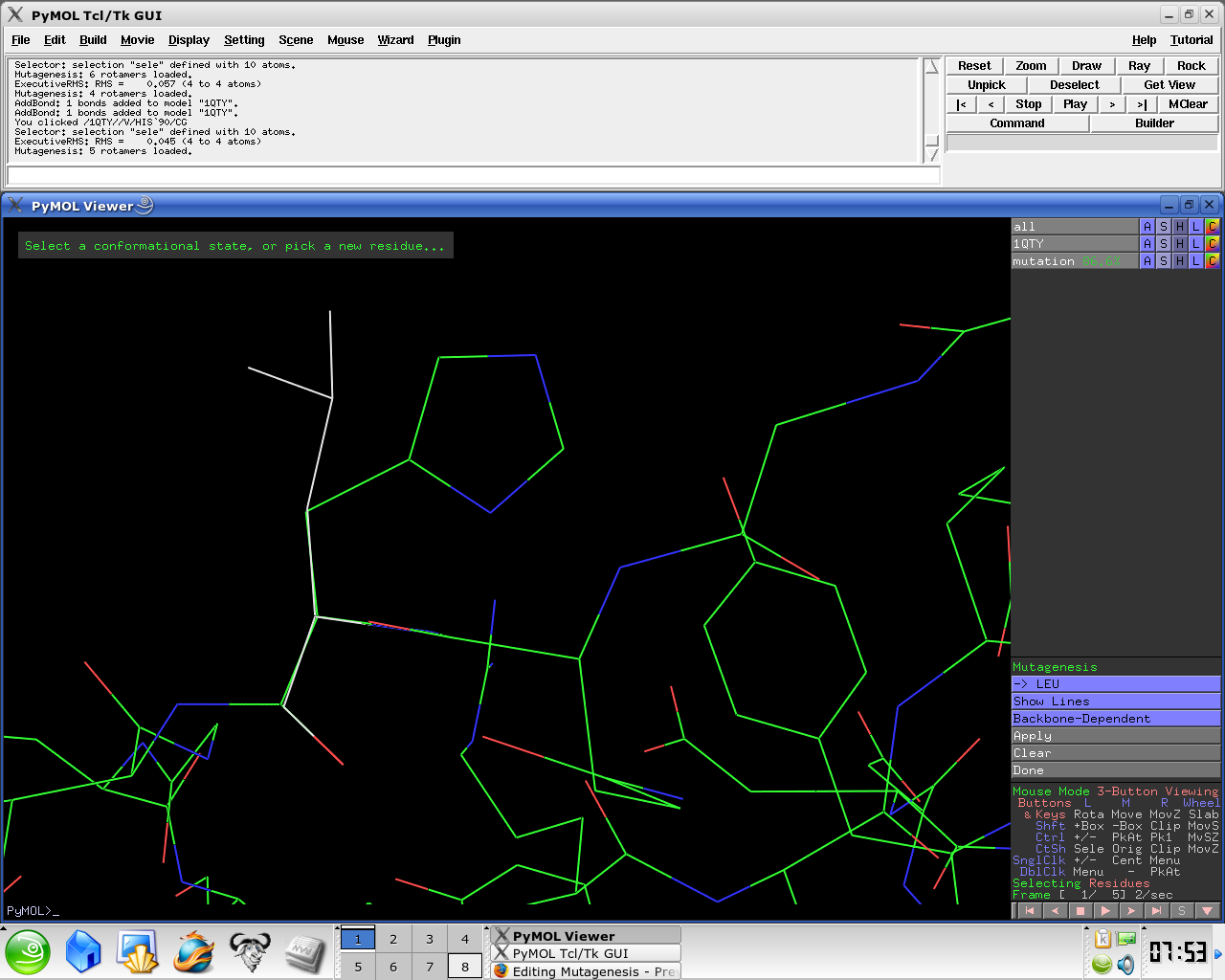
- 右下の進むと戻るのボタンをクリックして、rotamerを変更して立体障害の少ないものを選びます。
(赤や橙の円盤が立体障害です。)
- 右下の進むと戻るのボタンをクリックして、rotamerを変更して立体障害の少ないものを選びます。

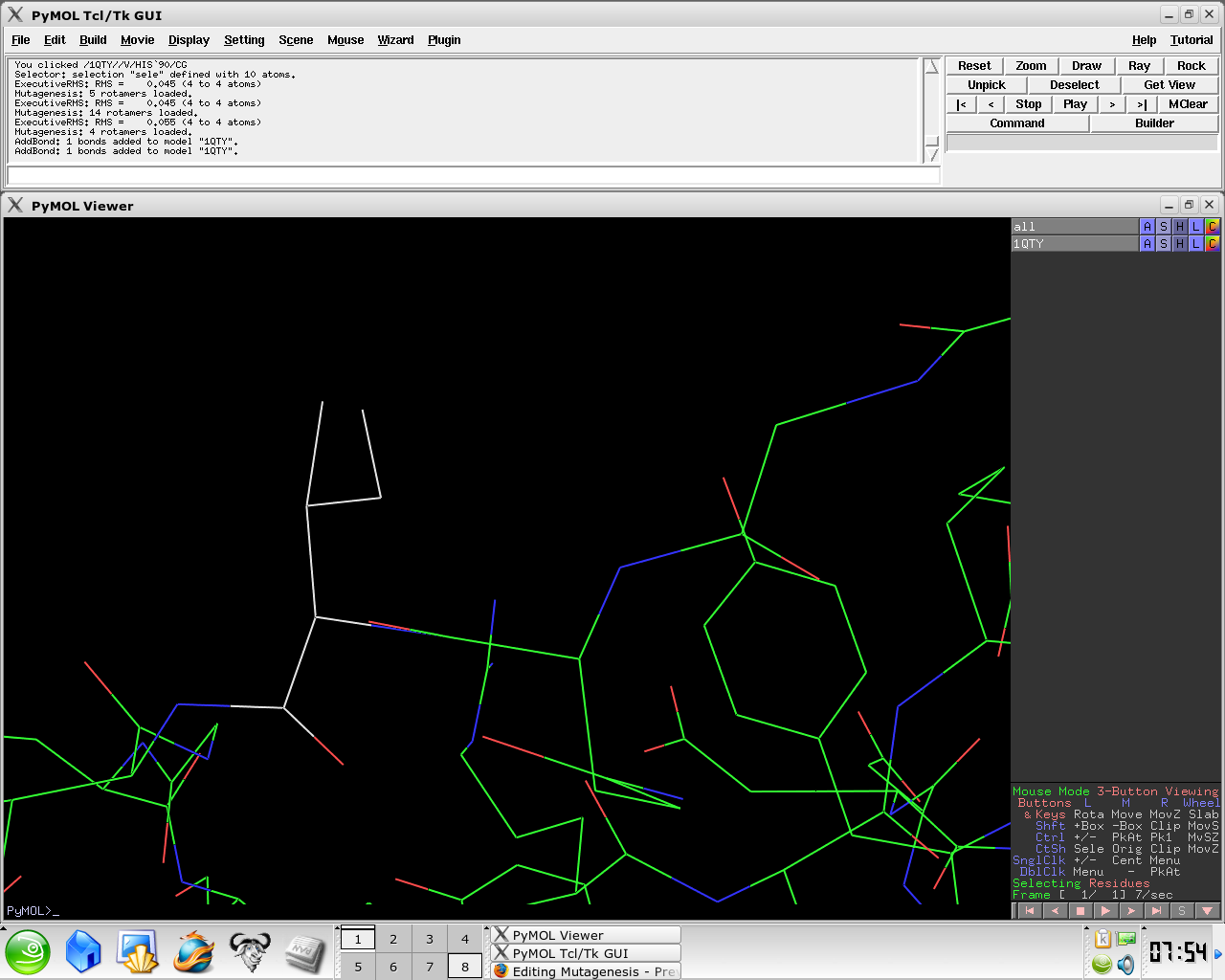
- Apply → Done の順でクリックして完了


PyMOLを使って正常な蛋白に変異を作り、その構造を解析することができます。PyMOLには、変異導入を簡単に行うための「Mutagenesis Wizard」があります。このウィザードを使うと、以下の手順で変異を導入できます:
PDBファイルをロードする。
ウィザードメニューから「Mutagenesis」を選択する。
PyMOLビューアウィンドウで変異を導入したい残基を選択する。
「No Mutation」を選択し、結果として得られる残基を選択する。
適切なロタマー(側鎖の立体配置)を選択する。
「Apply」を選択し、最後に「Done」を選択する。
これにより、蛋白質の構造に変異を導入することができます。さらに、PyMOLを使って変異後の蛋白質の構造解析を行うことも可能です。例えば、静電ポテンシャルの計算や分子動力学シミュレーションなどを行うことで、変異が蛋白質の機能や安定性に与える影響を評価することができます。
詳細な手順やチュートリアルについては、PyMOLWikiやおうちで解析のサイトを参考にしてください。
Mutagenesis
Mutagenesis
PyMol has a Mutagenesis Wizard to make mutagenesis very easy for the end user.
In rotkit, a function has been made to call a mutagenesis.
As of PyMOL version 2.2, users may now perform base mutations in nucleotide chains.
Walk-through
To mutate a residue follow these easy steps:
- Load a PDB file
- Under the Wizard menu select Mutagenesis
- In the PyMol viewer window select a residue
- Select No Mutation and select resultant residue
- Selecting the rotamer you think better fits your structure.
Several side chain orientations (rotamers) are possible. You can use the back-and-forth movie controls (lower right corner) to display (in white) each of the rotamers available for this residue in PyMOL, whose current and total numbers are shown in the (green) Frame info. The rotamers are ordered according to their frequencies of occurrence in proteins, shown as a red percentage at the mutation object, which exists while mutagenesis is being performed. - Select Apply
- Select Done
保存
File → Save sessionで保存することができます。
変異後に静電ポテンシャルなどを計算するとき(静電ポテンシャルの表示参照)は、
File → Save molecule
からPDBを選択します。
2024年11月9日 | カテゴリー:AUTODOCK VINA |