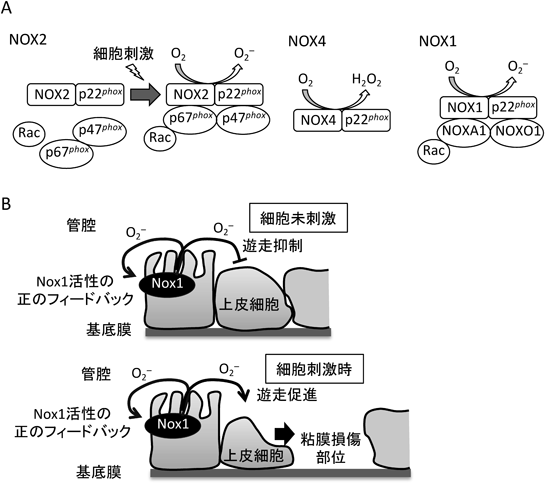
細胞REDOX機構
細胞内のレドックス(酸化還元)機構にはいくつかの重要なシステムがあります。以下に代表的な5種類を紹介します:
グルタチオンシステム:グルタチオンは細胞内で主要な抗酸化物質として機能し、グルタチオンペルオキシダーゼやグルタチオンリダクターゼと協力して過酸化水素などの活性酸素種を無害化します1。
チオレドキシンシステム:チオレドキシンは、酸化ストレス下でタンパク質のジスルフィド結合を還元する役割を持ち、チオレドキシンリダクターゼによって再生されます2。
スーパーオキシドジスムターゼ(SOD):SODはスーパーオキシドアニオンを過酸化水素に変換する酵素で、ミトコンドリアや細胞質に存在します1。
ペルオキシレドキシン:ペルオキシレドキシンは過酸化水素や有機過酸化物を還元する酵素で、細胞内の酸化ストレス応答に重要な役割を果たします2。
これらのシステムが協力して、細胞内の酸化還元バランスを維持し、酸化ストレスから細胞を保護しています。
タンパク質品質管理を支える小胞体レドックス環境と電子伝達
京都産業大学生命科学部/京都産業大学タンパク質動態研究所 ◇ 〒603–8555 京都市北区上賀茂本山
小胞体は分泌タンパク質や膜タンパク質のフォールディングの場として働く.タンパク質の立体構造形成に重要な翻訳後修飾であるジスルフィド結合形成を行うため,小胞体は酸化的レドックス環境を維持し,20種類を超える酸化還元酵素が存在する.このジスルフィド結合形成システムは,Ero1–PDI複合体を中心とする電子伝達経路によって,小胞体に挿入された新生ポリペプチド鎖からの電子を酸素へと受け渡すことで,ジスルフィド結合を導入する.また,酸化還元酵素が互いにネットワークを形成することで,多様な基質を効率よく酸化する.新たな計測技術の登場とともに,より詳細な小胞体での電子ネットワークの全体像が明らかになりつつある.本稿では,小胞体のレドックス環境を背景に織りなす生命の巧みなタンパク質品質管理機構について紹介したい.
© 2021 公益社団法人日本生化学会
リボソームで合成されたポリペプチド鎖は,それぞれ適切な立体構造を獲得して,はじめてタンパク質として機能を発揮することができる.細胞内では,分子シャペロンの働きによって,より正確に,より効率的に構造形成が成立する必要がある.分泌・膜タンパク質は,小胞体へ移行し,その多くがジスルフィド結合形成を伴う酸化的フォールディングを受ける.システイン残基の硫黄原子を介したジスルフィド結合は,タンパク質構造の安定性やサブユニット間の適切な配置に必要であり,その形成はタンパク質の機能発現に重要である.真核生物において,ジスルフィド結合形成を触媒する酵素としてprotein disulfide isomerase(PDI)とそのファミリータンパク質が知られており,PDIを再酸化する酵素としてER oxidoreductin 1(Ero1)が存在する.PDIは新生ポリペプチド鎖の硫黄原子から電子を授受することで酸化的フォールディングを触媒し,奪った電子はPDIからEro1へ,そしてEro1が持つflavin adenine dinucleotide(FAD)を介して分子状酸素に受け渡される.この電子伝達によって,小胞体内腔の酸化的フォールディングは成立するが,近年,PDIを含めた約20種類もの酸化還元酵素群が電子を伝達するネットワークを形成し,効率的な酸化的フォールディングを実現していることが明らかになった.さらに,PDI酸化反応を触媒する酵素が相次いで同定され,同じ真核生物でも酵母などと比べ,より複雑な酸化的フォールディングを支える分子基盤が明らかになりつつある.また,タンパク質の酸化還元状態をPDIファミリータンパク質が変えることで,ダイナミックな構造変化を引き起こし,レドックスが酵素の活性を制御する分子スイッチとして機能する例も相次いで明らかになっており,細胞内のシグナル伝達経路の一つとしても注目される.本稿では,小胞体を中心として,タンパク質構造形成を支える小胞体の酸化還元(レドックス)環境と電子伝達について解説する.
2. 小胞体のレドックス環境を理解する
1)細胞内で働くレドックス因子の酸化還元電位
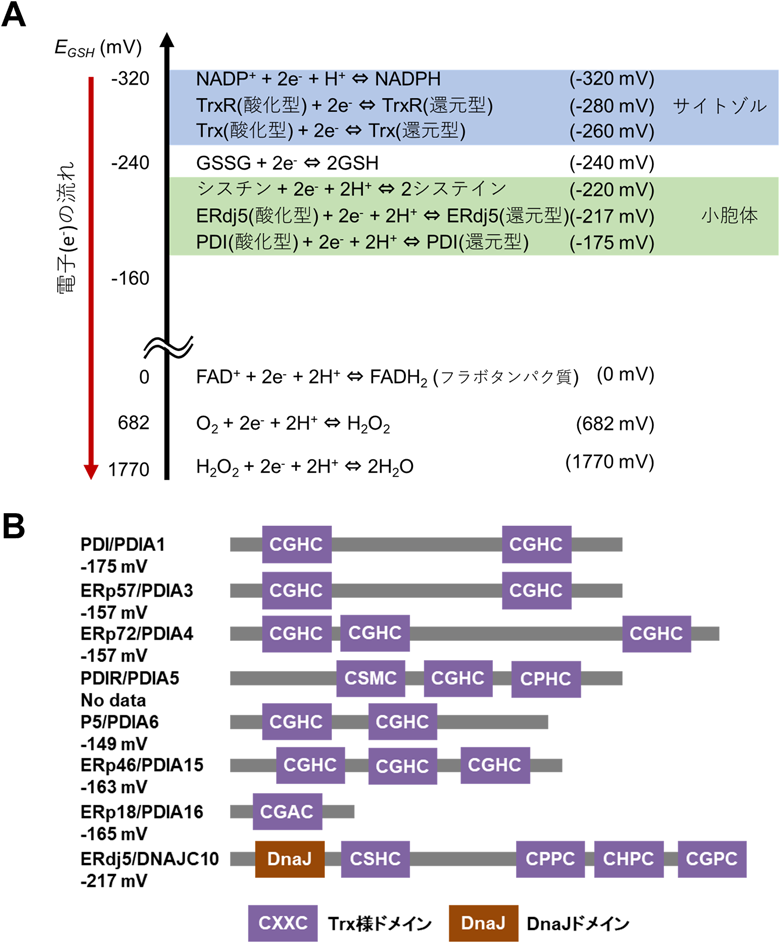
化学反応として酸化還元反応が実際に起こるかどうかを判断するうえで,重要な指標として酸化還元電位(レドックスポテンシャル)がある.生体物質において標準酸化還元電位を直接,決定することは容易ではなく,仲介分子を系に導入し,電位を測定した後に水素電極電位に換算することが一般的である.したがって,相対酸化還元電位[グルタチオン緩衝系など,細胞小器官(オルガネラ)環境に対応する酸化還元物質緩衝系]に基づく議論となるが,代表的な酸化還元物質については古くから酸化還元電位が決定されている(図1A)1).負の電位がポテンシャルエネルギーとして高い状態にあり,自発的な電子の移動は負から正の方向にのみ起こる.したがって,図1より,サイトゾルにおいて還元反応に関与するチオレドキシン(thioredoxin:Trx)とその還元酵素Trx reductase(TrxR),またその補因子であるnicotinamide adenine dinucleotide phosphate(NADPH)は,レドックスポテンシャルに従って,NADPHからTrxへと電子が移動する.また,システインとシスチンの酸化還元電位を例にとると,グルタチオンと比較しても,酸化的な電位をもつ.つまり,サイトゾルでは,NADPHの還元力をもとにTrxを介した,ジスルフィド結合の還元が起こりやすい.また,グルタチオンによっても,ジスルフィド結合の開裂が起こる可能性が高いことがわかる.このようにレドックスポテンシャルに従い,サイトゾル環境ではジスルフィド結合の形成が起こりにくいといえる.一方で,小胞体内腔では,ジスルフィド結合形成が自発的に進みやすく,さらに,この反応でPDIファミリータンパク質が電子を受け取るが,PDIファミリータンパク質の酸化還元電位に注目すると(図1B),還元酵素ERdj5を例外として,PDIファミリータンパク質はいずれもシステインよりも高いレドックスポテンシャルを持つことから2),基質からPDIファミリータンパク質へ電子伝達が起こることが理解できる.
図1 細胞内の酸化還元物質のレドックスポテンシャル
(A)細胞内の代表的な酸化還元物質の半反応式と酸化還元電位(mV)を示す.酸化還元反応において,電子は電位の低い方から高い方へ移動する.また,細胞内のレドックス環境は,グルタチオン平衡系をもとに,サイトゾルが約−300 mVという還元的環境に換算され,小胞体は約−200 mVというサイトゾルと比較して酸化的な環境となっている.(B)酸化還元活性が報告されているPDIファミリータンパク質の一例.それぞれのタンパク質名/遺伝子名と酸化還元電位およびドメイン構造を示す.PDIファミリータンパク質は,CXXCからなる活性中心モチーフ含むTrx様ドメインを分子内に持つ.ERdj5は,Hsp70結合領域であるDnaJドメインを持つ.
2)小胞体の酸化的環境とは
グルタチオンは比較的高濃度(0.1~10 mM)に存在するチオール分子である.細胞内のレドックス環境は,還元型グルタチオン(GSH)と酸化型グルタチオン(GSSG)によって緩衝され,その酸化還元比([GSH]2/[GSSG])に基づいて,酸化還元電位を考える.サイトゾルではGSHの割合が高く([GSH]:[GSSG]で,およそ100:1),還元的な環境とされる.このような環境下では,ジスルフィド結合が形成されることはほとんどない.対して,小胞体はGSSGの比率が高く([GSH]:[GSSG]で,おおよそ1~10:1),相対的に酸化的な環境といえる.以前より,生化学的な手法によってGSHの量が測定され,細胞内のGSHの濃度や酸化還元比が見積もられてきた.しかし,生細胞内で正確にGSH濃度,特にオルガネラ内のGSH濃度を測定することは困難であった.GFP改変型のレドックスセンサータンパク質roGFPの開発と近年の改良によって,生細胞内においてレドックス環境の可視化が実現され,生細胞内のGSH濃度の測定が可能になった3).驚くべきことに,小胞体の総量としてのGSH濃度は15 mMとサイトゾル全体の値(7 mM)よりも高く,小胞体膜を隔てたサイトゾルとで濃度勾配が生じることが示唆されている4).小胞体のレドックスポテンシャルはいまだに決定的な値は定かではないが,およそ−200 mV程度と考えられている.このような環境下において,GSHやシステインがPDIファミリータンパク質によって酸化されやすいことが説明でき,PDIからEro1への電子伝達もレドックスポテンシャルから考えると進行しやすい反応であることがわかる(詳細は後述).ただし,GSSGが多く存在するということが,直接的にジスルフィド結合形成を触媒するということではない.小胞体のレドックスポテンシャルが約−200 mVという環境のもとで平衡状態を考えたとき,チオール基として存在するよりも,ジスルフィド結合を形成した方が安定であり,その存在比が多くなるということである.また,酸化的なレドックスポテンシャルをもつPDIファミリータンパク質の活性中心は,ほとんどが開裂した還元型として存在することになり,常にドナーから電子を供給されることになる(つまり酸化酵素として機能する).ERdj5は酸化型が多く存在することになり,常にアクセプターに対して電子を与えることになる(つまり還元酵素として機能する).また,GSHは活性酸素種(reactive oxygen species:ROS)と反応して,酸化型へと変換される.したがって,サイトゾルの環境とは対照的に,グルタチオン平衡系におけるGSSGの比率が大きくなることで,システイン残基は必然的に還元されにくく,かつPDIファミリータンパク質によって絶えず酸化される環境にある.このことが,小胞体を酸化的フォールディングの場とし,ジスルフィド結合形成が小胞体で優位に進む理由である.
3. 真核生物におけるジスルフィド結合形成システム
1)Ero1–PDI複合体を中心とする電子伝達経路
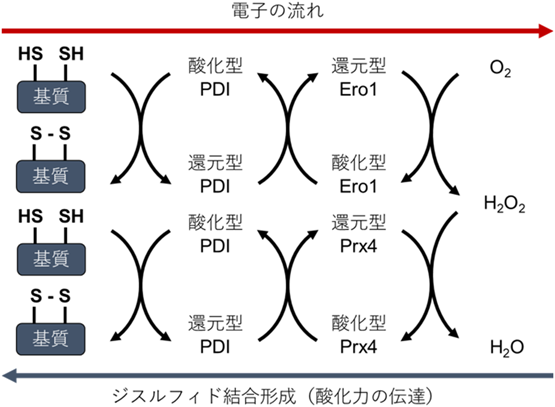
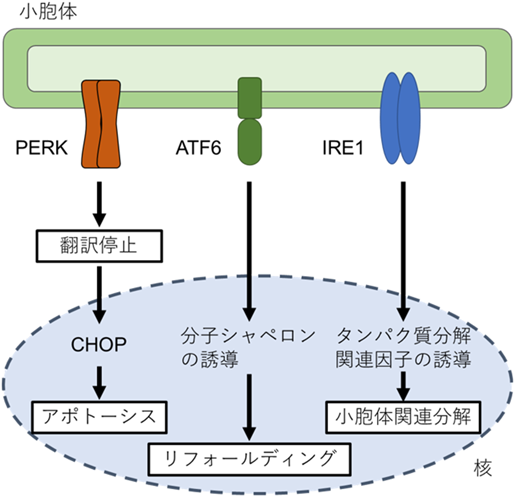
小胞体に挿入された分泌・膜タンパク質は,小胞体で酸化的フォールディングを介して成熟する.小胞体でタンパク質のフォールディングを触媒する酵素の存在が発見されたのは1963年のことであり,StraubらとAnfinsenらのグループがそれぞれ独立して発見した5, 6).この酵素がPDIであり,その後,遺伝子クローニングにより決定された塩基配列から,分子内に相同性の高い領域が存在することがわかった.この相同性の高い領域は,大腸菌のチオレドキシンと類似しており,チオレドキシン様ドメインと名づけられた.チオレドキシン様ドメインは,ドメイン内に活性中心モチーフCXXCを持つ7).AnfinsenはPDIをジスルフィド結合の異性化酵素と考え,精製したリコンビナントPDIは,in vitroで高い異性化活性を示した8).この時点で,小胞体が他のオルガネラと比較して,酸化的な環境であることはわかっていたが,まだジスルフィド結合形成メカニズムの詳細は明らかではなかった9).PDIの発見から遅れること約40年,1998年にWeissmanとKaiserのラボにおいて独立にPDIを酸化する酵素Ero1が酵母で同定された10, 11).翌年,Ero1の酵母ホモログEro1pがPDIを酸化し,PDIの活性中心にジスルフィド結合を形成することが明らかとなり,PDIが異性化活性だけでなく,酸化酵素として基質のジスルフィド結合形成反応に大きく寄与することが明らかとなった12).その後,ヒトEro1は2000年にSitiaらが同定し13, 14),小胞体に挿入された新生ポリペプチド鎖のシステイン残基から電子がPDIに受け渡され,Ero1へと伝達されることが明らかになった.この電子伝達によって,新生ポリペプチド鎖にジスルフィド結合が形成され,電子を受け取ったPDIは,Ero1によって再酸化される.Ero1は活性中心にFADが結合したフラボタンパク質であり,Ero1がPDIを酸化するとき,Ero1はPDIから電子を二つ受け取る.この二つの電子はFADを介して分子状酸素(O2)へと受け渡され,還元された酸素は過酸化水素(H2O2)へと変換される(図2).ROSの一種である過酸化水素は酸化ストレスの要因ともなりうるが,過剰に過酸化水素が産生されないように,Ero1自身の活性をシステイン残基間のジスルフィド結合を架け替えることで制御するフィードバック機構が存在する15).Ero1は酵母からヒトまで真核生物で進化的に保存されている.酵母ではEro1pと1種類であるが,ヒトではEro1αとEro1βの2種類が存在する.Ero1αは広範な組織で発現が認められるのに対し,Ero1βは膵臓など分泌タンパク質が多く合成される組織で高い発現が認められている.このEro1αとEro1βの発現パターンの違いには,両者の転写因子の違いに起因するといわれている.小胞体内腔にフォールディング不全タンパク質が蓄積すると,いくつかの小胞体ストレスセンサータンパク質の活性化に依存して,転写因子が働き,分子シャペロンや酸化還元酵素群などタンパク質品質管理に関わる因子を誘導し,恒常性を維持する(図3).Ero1αはCHOPによって誘導される遺伝子であり,Ero1βはATF6下流の遺伝子として小胞体ストレスによって誘導される.分泌組織の細胞はタンパク質合成が盛んであり,常に小胞体ストレスが惹起された状態と言っても過言ではない16).Ero1βがATF6下流に存在し,分泌組織で高発現が認められることは,分泌細胞の機能と合目的的な役割が推察できる.酵母ではEro1pを欠損するとタンパク質の酸化的フォールディングが進まず,小胞体ストレスが生じ,酵母は死んでしまう.しかし,哺乳類における2種類のEro1αとEro1βの二重欠損マウスは正常に発生し,生育可能である.驚くべきことに,タンパク質の酸化的フォールディングにも異常が見られず,小胞体の酸化的環境も維持される17).この結果から,哺乳類ではEro1以外のPDIファミリータンパク質への酸化酵素の存在が示唆され,酸化的フォールディングシステムの補完性が考えられる.
図2 小胞体におけるEro1とPrx4による電子伝達
還元状態にある基質のチオール基(–SH)は,PDIによって酸化され,2電子をPDIに渡す.還元型PDIはEro1に電子を渡し,Ero1はFADを介して分子状酸素(O2)を還元し,過酸化水素(H2O2)が発生する.H2O2はPrx4によって水(H2O)へと還元され,その酸化力はPrx4を介して,再びPDIファミリータンパク質へと伝達する.
図3 小胞体ストレス応答
小胞体に不良タンパク質が蓄積することで惹起される転写誘導を小胞体ストレス応答と呼ぶ.小胞体ストレス応答では,PERK・ATF6・IRE1の三つがストレスセンサーとして不良タンパク質の蓄積を感知し,翻訳の一時停止,分子シャペロンの発現誘導,タンパク質分解関連因子の誘導,そしてアポトーシスが引き起こされる.これらセンサータンパク質も一部レドックス制御が関わることが知られ,レドックス環境の変化は小胞体内腔のタンパク質恒常性機構をダイナミックに変化させる可能性がある.
2)小胞体内の電子伝達の全体像
Ero1の発見以降,peroxiredoxin 4(Prx4),vitamin K epoxide reductase(VKOR),glutathione peroxidase 7(Gpx7)およびGpx8の4種類の酸化酵素が相次いで報告され,NADPH oxidase 4(Nox4)も小胞体膜上で酸化酵素として機能することが報告されている18).還元型Prx4は還元型PDIから受け取った電子を過酸化水素(H2O2)へと受け渡し,過酸化水素をさらに水(H2O)に還元する反応を触媒する(図2).このように細胞内に過剰な過酸化水素が蓄積しないように,また効率よく電子を処理する仕組みを備えている19).Prx4の遺伝子欠損マウスも作製されたが,前述のEro1α/β欠損マウスと同様に,正常に生育し20),タンパク質のフォールディングにも異常が認められなかった.ところが,Ero1αとβの二重欠損マウスにさらにPrx4を遺伝子欠損することによって顕著にマウスの出生率が低下することから,Ero1とPrx4の両酸化的フォールディングシステムが協調的に働くことが,初期発生において重要であると示唆される21).Gpx7/8はグルタチオンペルオキシダーゼのファミリーとして分類されているが,他のGpxファミリーが活性中心にセレノシステインを持ちGSHの還元力を用いて基質を還元するのに対して,Gpx7/8は通常のシステインを活性中心に持ち,過酸化水素を用いてPDIファミリータンパク質の酸化に寄与する22).Nox4はNoxファミリーに分類される酵素であり,酸素を電子受容体としてNADPHの酸化を触媒し,小胞体内にROSを産生させることによって小胞体の酸化的環境の維持に寄与することが知られている23).PDIと相互作用することから,直接的な電子の授受も示唆されている24).Ero1がPDIに比較的高い特異性をもってジスルフィド結合形成を促進するのに対して,これらの酸化酵素はPDIファミリータンパク質全般に対して幅広く作用していることが報告されている25).また,Erv2[酵母にのみ存在,ヒトの相同タンパク質はquiescin sulfhydryl oxidase(QSOX)である]やER flavoprotein associated with degradation(ERFAD)といった新たなフラボタンパク質の酸化還元反応の生物学的意義は明らかになっていないが,小胞体に局在することが明らかになっている26, 27).現在では,PDI–Ero1経路の発見当初の予想よりもはるかに複雑なジスルフィド結合形成のネットワークの存在が考えられている.
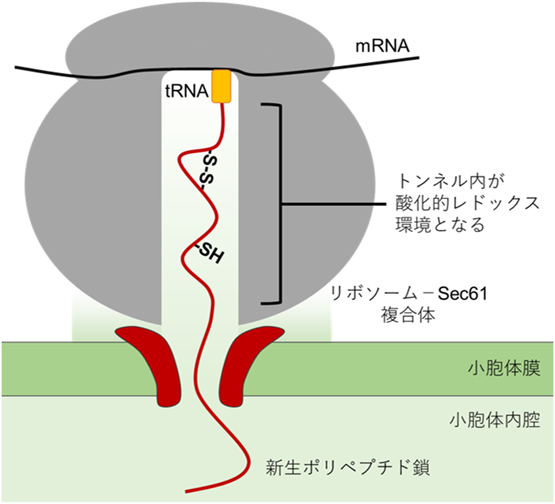
3)新生ポリペプチド鎖へのジスルフィド結合導入の新展開
ここまで小胞体内腔での酸化的フォールディングと電子伝達について論じてきたが,近年,翻訳と共役した新生ポリペプチド鎖のジスルフィド結合形成について,興味深い知見が次々と明らかになった.タンパク質の翻訳に共役して分泌タンパク質はSecトランスロコンチャネルを通過するが,リボソームとSecが結合した段階で,リボソームトンネル内の環境がサイトゾルの還元的環境から小胞体の酸化的な環境へシフトし(図4),リボソームトンネル内で自発的なジスルフィド結合形成が起こる可能性が示唆された28).ただし,リボソームトンネル内の大きさは狭く,ポリペプチド鎖の立体構造形成はαヘリックス構造といった単純なものであり,また近接するアミノ酸残基間における酸化反応のみに限られる.また,小胞体内腔に露出した新生ポリペプチド鎖の初期段階でのジスルフィド結合形成には,積極的にERp46が関与するとの報告がある29).ERp46はPDIファミリータンパク質の中では酸化酵素としての活性が強く,非選択的にポリペプチド中のシステイン残基を酸化し,ジスルフィド結合形成を促進していると考えられている.また,PDIはシャペロン活性も持ち合わせており,ジスルフィド結合の異性化酵素活性も高いことから,新生鎖にランダムに導入されたジスルフィド結合を天然構造型のジスルフィド結合へと校正していくメカニズムが提唱されている.このように,新生ポリペプチドのステップワイズなジスルフィド結合形成とPDIファミリーの関わりについて詳細が明らかになりつつある.
4. タンパク質品質管理機構とPDIファミリータンパク質
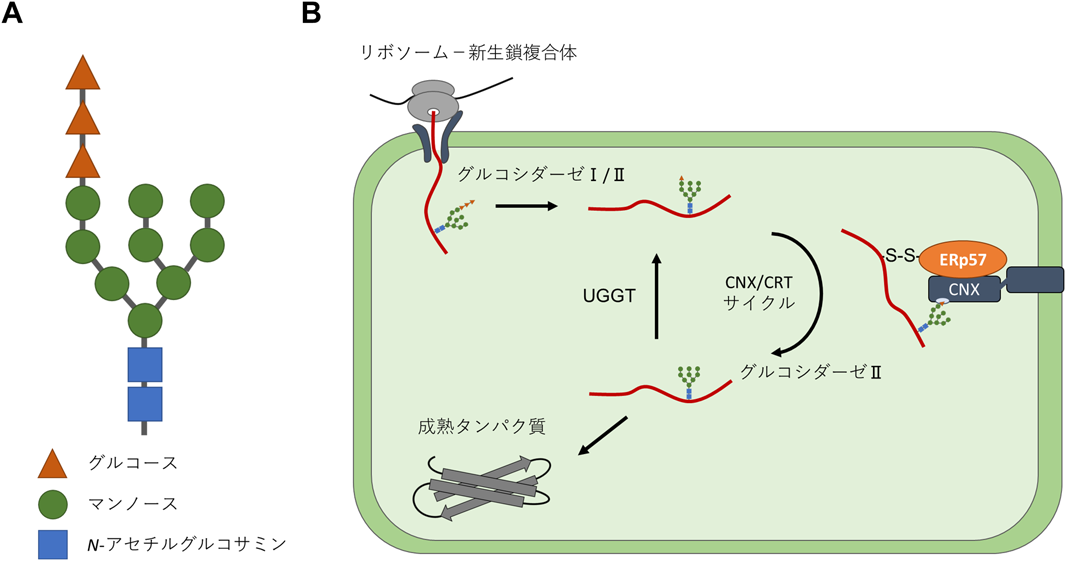
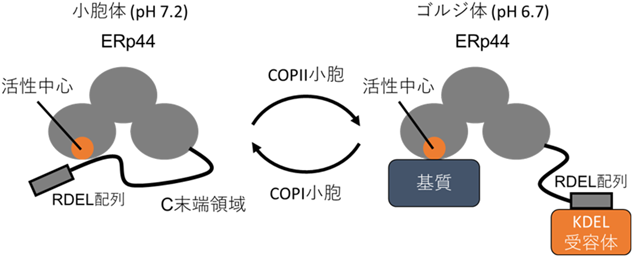
小胞体に挿入される分泌タンパク質の大半は糖タンパク質であり,翻訳に共役して小胞体でN型糖鎖修飾を受ける.小胞体に滞留する糖タンパク質の糖鎖構造は,小胞体のグルコシダーゼとマンノシダーゼによってトリミングされる.グルコシダーゼI, IIによって順次グルコースがトリミングされ,モノグルコース型となった糖鎖を持つ糖タンパク質は,レクチン様活性を持つ分子シャペロンCalnexin(CNX)またはCalreticulin(CRT)に認識され,糖タンパク質のフォールディングを促進する30).また,CNXとCRTは,PDIファミリータンパク質であるERp57やERp29との結合領域をもつ.ERp57はCNXあるいはCRTを足場として,基質のジスルフィド結合形成を触媒する.ERp57はPDIと異なり,基質と直接的に相互作用するために必要な疎水性相互作用領域を持たないが,CNX/CRTと協調することで,多様な基質のジスルフィド結合形成に寄与している.ここで正しい立体構造を獲得したタンパク質は,さらにグルコースがトリミングされ,CNXおよびCRTから解離し,ゴルジ体へと輸送される.フォールディングが不完全な場合には,フォールディングセンサーとして機能するUDP-glucose:glycoprotein glucosyltransferase(UGGT)によってグルコースが再付加され,再びCNXおよびCRTによって認識され,フォールディングが再び試みられる.このような糖タンパク質の品質管理機構をCNX/CRTサイクルと呼び,糖鎖構造を“目印”とするフォールディング監視機構が詳細に解明されてきた(図5).このようなCNX/CRTのようなレクチン型シャペロンによる厳密な品質管理にもかかわらず,この品質管理をかいくぐり,フォールディング不全タンパク質が分泌経路に進んでしまった場合,PDIファミリータンパク質であるERp44が小胞体へと再び基質を引き戻し,小胞体で再度フォールディングが試みられる.ERp44は小胞体–ゴルジ中間体に局在するPDIファミリータンパク質であり,酸化還元酵素としての活性を持ち合わせていないが,基質分子と活性中心のシステイン残基を介して分子間ジスルフィド結合を形成することができる31).また,ERp44はカルボキシ末端に小胞体残留シグナルを持つため,KDEL受容体の働きによってERp44–基質複合体として小胞体へと逆輸送される32).このERp44のフォールディング監視機構は,ERp44の活性モチーフに対して,フレキシブルな構造を持つカルボキシ末端領域が覆いかぶさる構造をとるか否かがpHに依存している点に特徴があり33),小胞体の中性条件とシスゴルジの弱酸性環境の違いを巧みに利用している(図6).ERp44の代表的な基質としては,免疫応答の最初に産生される免疫グロブリンMや脂肪細胞から分泌されるアディポサイトカインの一種であるアディポネクチンなどの分泌タンパク質が知られている34).これらは成熟の過程でジスルフィド結合依存的なオリゴマーを形成する.免疫グロブリンMは,最初に産生される抗体でありながら,五量体という免疫グロブリンのサブクラスの中では最も大きな構造をとるタンパク質である.抗原に対する特異的抗体の産生を活性化する古典的経路は,免疫グロブリンMと補体の結合から開始するため,小胞体におけるタンパク質品質管理が重要となる.また,アディポネクチンは,インスリン抵抗性の改善や動脈硬化の防止作用など,生体に有益な生理活性物質として注目されている.これらのさまざまな有益な効果は,三量体を基本単位として多量体化したアディポネクチンによって発揮されるため,やはりタンパク質フォールディングの厳密な品質管理が重要である.ERp44はそのようなオリゴマー化に関わるチオール基とジスルフィド結合を形成することで,未成熟なまま分泌経路へ入ってしまったフォールディング中間体の基質を小胞体へと逆輸送する.また,血圧調節に重要なアンジオテンシンの分解酵素であるERAP1とジスルフィド結合依存的な複合体を形成することで,ERAP1を細胞内にとどめる働きがあると報告された35).ERAP1の分泌量を調節することによって,血圧の制御を行っていると考えられる.このフォールディング監視機構には,分泌タンパク質の量的制御の役割もあるといえる.このように未成熟タンパク質が分泌経路に入らないようにする仕組み,もし未成熟なタンパク質が分泌経路に入ってしまってもそれを監視し,小胞体へと再び戻す仕組みという二重の品質管理機構を備え,厳密性を担保している.
図5 糖鎖構造に依存したタンパク質フォールディング監視機構
(A) N型糖鎖のコア構造.新生鎖が小胞体に挿入されると,N型糖鎖付加部位のアスパラギン側鎖に付加される.(B)糖鎖構造に依存した糖タンパク質の品質管理.小胞体に存在するグルコシダーゼおよびマンノシダーゼによって糖鎖トリミングが進む.CNXおよびCRTはモノグルコース構造を認識し,ERp57と協調してフォールディングを介助する.グルコシダーゼIIによってグルコースがトリミングされるとCNX/CRTから解離し,フォールディングが不完全であればUGGTによってグルコースの再付加が起こる.成熟したタンパク質はゴルジ体へ輸送される.
5. PDIファミリータンパク質の補完性と多様な機能
このように小胞体には多くの酸化還元酵素が存在している.これは多細胞生物に進化し,細胞間コミュニケーションの必要性が生まれることで,多種多様な分泌・膜タンパク質の立体構造形成に関与する必要が生じたと考えられるが,その理由は明確ではない.酵母においてはEro1の欠損が致死であるのに対して,動物細胞では影響が観察されなかったことは前述したが,同様にPDIも酵母においては欠損が致死であるのに対して36),動物細胞では重篤な異常はみられない.さらに約20種類存在するPDIファミリータンパク質のうち遺伝子欠損マウスで致死となることが報告されているものは,ERp57のみである37).したがって,ERp57が初期発生に必須であることは明らかであるが,培養細胞レベルではERp57欠損細胞も生育可能である.特異的な機能をもつ酸化還元酵素がある一方で,細胞のロバストネスを担保するための酵素の存在が予想される.細胞の生死を判断基準とすると,一つの酸化還元酵素が機能不全に陥ったとしても極端な影響はみられないが,特定のタンパク質を過剰発現させるなどの負荷を与えたときには,顕著に脆弱性を示すこともある38).それぞれのPDIファミリータンパク質の基質特異性についても精力的に研究が続けられているが39),個々の機能解析が進んでいる遺伝子もあれば,酵素活性の有無すら報告されていないものも多く,今後の解明が待たれる.
ここまでPDIファミリータンパク質による酸化的フォールディングに焦点を当て,論じたが,近年,PDIファミリータンパク質による酸化還元反応はフォールディングのみに限らず,転写因子やイオンポンプ,イオンチャネルといった生体内で重要な役割を果たすタンパク質の活性制御に関わることが明らかになってきた40–42).これまでに我々のグループでは,小胞体のカルシウムポンプであるSERCA2bのポンプ活性を上昇させる因子として還元酵素ERdj5を報告している43, 44).また,PDIRやERp18は小胞体ストレスセンサーATF6の制御に関わり45, 46),P5はもう一つの小胞体ストレスセンサーIRE1の制御に寄与する47)という酸化還元酵素によるERストレスセンサーの制御が報告されており,小胞体の酸化還元環境が細胞全体の恒常性維持に密接に関わっていることが明らかになりつつある.
6. おわりに;今後の展望
PDIの発見以降,酸化的フォールディングの理解は飛躍的に進み,新生ポリペプチド鎖からPDI, Ero1そして分子状酸素へと直線的な電子伝達が示されてきた.しかし,近年,小胞体内腔での電子伝達は,硫黄原子を媒体としてネットワークを組み,複雑であることがわかってきた.このネットワーク形成が,小胞体内腔の恒常性に頑強性をもたらすものであり,また,より複雑なタンパク質高次構造体形成を可能にしているかもしれない.生体内の酸化的フォールディングをより理解するためには,小胞体での硫黄を媒体とした電子ネットワーク形成の解明が必須である.しかし,酸化的環境下での電子伝達がどのように効率よく成立するのかはいまだ不明である.また,細胞内にありながら,小胞体内腔がサイトゾルと比較して,どのように酸化的に維持されているのか,まったくわかっていない.超硫黄分子・修飾の実態が明らかにされ,これまで常識では考えられなかった電子伝達経路や,タンパク質構造形成が明らかにされるかもしれない.これら新たな知見が小胞体レドックス環境の実態解明につながることが大いに期待される.
引用文献
1) Xiao, W. & Loscalzo, J. (2020) Metabolic responses to reductive stress. Antioxid. Redox Signal., 32, 1330–1347.
2) Araki, K., Shun-ichiro, I., Kamiya, Y., Ron, D., Kato, K., Natsume, T., & Nagata, K. (2013) Ero1-α and PDIs constitute a hierarchical electron transfer network of endoplasmic reticulum oxidoreductases. J. Cell Biol., 202, 861–874.
3) Bilan, D.S. & Belousov, V.V. (2017) New tools for redox biology: From imaging to manipulation. Free Radic. Biol. Med., 109, 167–188.
4) Montero, D., Tachibana, C., Rahr Winther, J., & Appenzeller-Herzog, C. (2013) Intracellular glutathione pools are heterogeneously concentrated. Redox Biol., 1, 508–513.
5) Venetianer, P. & Straub, F.B. (1963) The enzymic reactivation of reduced ribonuclease. Biochim. Biophys. Acta, 67, 166–168.
6) Goldberger, R.F., Epstein, C.J., & Anfinsen, C.B. (1963) Acceleration of reactivation of reduced bovine pancreatic ribonuclease by a microsomal system from rat liver. J. Biol. Chem., 238, 628–635.
7) Freedman, R.B. (1984) Native disulphide bond formation in protein biosynthesis: Evidence for the role of protein disulphide isomerase. Trends Biochem. Sci., 9, 438–441.
8) Weissman, J.S. & Kimt, P.S. (1993) Efficient catalysis of disulphide bond rearrangements by protein disulphide isomerase. Nature, 365, 185–188.
9) Helenius, A., Marquardt, T., & Braakman, I. (1992) The endoplasmic reticulum as a protein-folding compartment. Trends Cell Biol., 2, 227–231.
10) Pollard, M.G., Travers, K.J., & Weissman, J.S. (1998) Ero1p: A novel and ubiquitous protein with an essential role in oxidative protein folding in the endoplasmic reticulum. Mol. Cell, 1, 171–182.
11) Frand, A.R. & Kaiser, C.A. (1998) The ERO1 gene of yeast is required for oxidation of protein dithiols in the endoplasmic reticulum. Mol. Cell, 1, 161–170.
12) Frand, A.R. & Kaiser, C.A. (1999) Ero1p oxidizes protein disulfide isomerase in a pathway for disulfide bond formation in the endoplasmic reticulum. Mol. Cell, 4, 469–477.
13) Cabibbo, A., Pagani, M., Fabbri, M., Rocchi, M., Farmery, M.R., Bulleid, N.J., & Sitia, R. (2000) ERO1-L, a human protein that favors disulfide bond formation in the endoplasmic reticulum. J. Biol. Chem., 275, 4827–4833.
14) Pagani, M., Fabbri, M., Benedetti, C., Fassio, A., Pilati, S., Bulleid, N.J., Cabibbo, A., & Sitia, R. (2000) Endoplasmic reticulum oxidoreductin 1-Lβ (ERO1-Lβ), a human gene induced in the course of the unfolded protein response. J. Biol. Chem., 275, 23685–23692.
15) Inaba, K., Masui, S., Iida, H., Vavassori, S., Sitia, R., & Suzuki, M. (2010) Crystal structures of human Ero1α reveal the mechanisms of regulated and targeted oxidation of PDI. EMBO J., 29, 3330–3343.
16) Iwawaki, T., Akai, R., Kohno, K., & Miura, M. (2004) A transgenic mouse model for monitoring endoplasmic reticulum stress. Nat. Med., 10, 98–102.
17) Zito, E., Chin, K., Blais, J., Harding, H.P., & Ron, D. (2010) ERO1-β, a pancreas-specific disulfide oxidase, promotes insulin biogenesis and glucose homeostasis. J. Cell Biol., 188, 821–832.
18) Wu, R.-F., Ma, Z., Liu, Z., & Terada, L.S. (2010) Nox4-derived H2O2 mediates endoplasmic reticulum signaling through local ras activation. Mol. Cell. Biol., 30, 3553–3568.
19) Zito, E., Melo, E.P., Yang, Y., Wahlander, Å., Neubert, T.A., & Ron, D. (2010) Oxidative protein folding by an endoplasmic reticulum-localized peroxiredoxin. Mol. Cell, 40, 787–797.
20) Iuchi, Y., Okada, F., Tsunoda, S., Kibe, N., Shirasawa, N., Ikawa, M., Okabe, M., Ikeda, Y., & Fujii, J. (2009) Peroxiredoxin 4 knockout results in elevated spermatogenic cell death via oxidative stress. Biochem. J., 419, 149–158.
21) Zito, E., Hansen, H.G., Yeo, G.S.H., Fujii, J., & Ron, D. (2012) Endoplasmic reticulum thiol oxidase deficiency leads to ascorbic acid depletion and noncanonical scurvy in mice. Mol. Cell, 48, 39–51.
22) Kanemura, S., Sofia, E.F., Hirai, N., Okumura, M., Kadokura, H., & Inaba, K. (2020) Characterization of the endoplasmic reticulum–resident peroxidases GPx7 and GPx8 shows the higher oxidative activity of GPx7 and its linkage to oxidative protein folding. J. Biol. Chem., 295, 12772–12785.
23) Takac, I., Schröder, K., Zhang, L., Lardy, B., Anilkumar, N., Lambeth, J.D., Shah, A.M., Morel, F., & Brandes, R.P. (2011) The E-loop is involved in hydrogen peroxide formation by the NADPH oxidase Nox4. J. Biol. Chem., 286, 13304–13313.
24) Janiszewski, M., Lopes, L.R., Carmo, A.O., Pedro, M.A., Brandes, R.P., Santos, C.X., & Laurindo, F.R. (2005) Regulation of NAD(P)H oxidase by associated protein disulfide isomerase in vascular smooth muscle cells. J. Biol. Chem., 280, 40813–40819.
25) Sato, Y., Kojima, R., Okumura, M., Hagiwara, M., Masui, S., Maegawa, K., Saiki, M., Horibe, T., Suzuki, M., & Inaba, K. (2013) Synergistic cooperation of PDI family members in peroxiredoxin 4-driven oxidative protein folding. Sci. Rep., 3, 2456.
26) Kodali, V.K. & Thorpe, C. (2010) Oxidative protein folding and the quiescin-sulfhydryl oxidase family of flavoproteins. Antioxid. Redox Signal., 13, 1217–1230.
27) Riemer, J., Appenzeller-Herzog, C., Johansson, L., Bodenmiller, B., Hartmann-Petersen, R., & Ellgaard, L. (2009) A luminal flavoprotein in endoplasmic reticulum-associated degradation. Proc. Natl. Acad. Sci. USA, 106, 14831–14836.
28) Schulte, L., Mao, J., Reitz, J., Sreeramulu, S., Kudlinzki, D., Hodirnau, V.V., Meier-Credo, J., Saxena, K., Buhr, F., Langer, J.D., et al. (2020) Cysteine oxidation and disulfide formation in the ribosomal exit tunnel. Nat. Commun., 11, 1–11.
29) Hirayama, C., Machida, K., Noi, K., Murakawa, T., Okumura, M., Ogura, T., Imataka, H., & Inaba, K. (2021) Distinct roles and actions of protein disulfide isomerase family enzymes in catalysis of nascent-chain disulfide bond formation. iScience, 24, 102296.
30) Araki, K. & Nagata, K. (2012) Protein folding and quality control in the ER. Cold Spring Harb. Perspect. Biol., 4, 1–25.
31) Anelli, T., Alessio, M., Mezghrani, A., Simmen, T., Talamo, F., Bachi, A., & Sitia, R. (2002) ERp44, a novel endoplasmic reticulum folding assistant of the thioredoxin family. EMBO J., 21, 835–844.
32) Kakihana, T., Araki, K., Vavassori, S., Iemura, S., Cortini, M., Fagioli, C., Natsume, T., Sitia, R., & Nagata, K. (2013) Dynamic regulation of Ero1α and peroxiredoxin 4 localization in the secretory pathway. J. Biol. Chem., 288, 29586–29594.
33) Vavassori, S., Cortini, M., Masui, S., Sannino, S., Anelli, T., Caserta, I.R., Fagioli, C., Mossuto, M.F., Fornili, A., van Anken, E., et al. (2013) A pH-regulated quality control cycle for surveillance of secretory protein assembly. Mol. Cell, 50, 783–792.
34) Cortini, M. & Sitia, R. (2010) From antibodies to adiponectin: Role of ERp44 in sizingand timing protein secretion. Diabetes Obes. Metab., 12(Suppl 2), 39–47.
35) Hisatsune, C., Ebisui, E., Usui, M., Ogawa, N., Suzuki, A., Mataga, N., Takahashi-Iwanaga, H., & Mikoshiba, K. (2015) ERp44 exerts redox-dependent control of blood pressure at the ER article ERp44 exerts redox-dependent control of blood pressure at the ER. Mol. Cell, 58, 1015–1027.
36) Nørgaard, P., Westphal, V., Tachibana, C., Alsøe, L., Holst, B., & Winther, J.R. (2001) Functional differences in yeast protein disulfide isomerases. J. Cell Biol., 153, 553–562.
37) Wang, Y., Nizkorodov, A., Riemenschneider, K., Lee, C.S., Olivares-Navarrete, R., Schwartz, Z., & Boyan, B.D. (2014) Impaired bone formation in pdia3 deficient mice. PLoS One, 9, 1–11.
38) Hosoda, A., Tokuda, M., Akai, R., Kohno, K., & Iwawaki, T. (2010) Positive contribution of ERdj5/JPDI to endoplasmic reticulum protein quality control in the salivary gland. Biochem. J., 425, 117–125.
39) Jessop, C.E., Watkins, R.H., Simmons, J.J., Tasab, M., & Bulleid, N.J. (2009) Protein disulphide isomerase family members show distinct substrate specificity: P5 is targeted to BiP client proteins. J. Cell Sci., 122, 4287–4295.
40) Brigelius-Flohé, R. & Flohé, L. (2011) Basic principles and emerging concepts in the redox control of transcription factors. Antioxid. Redox Signal., 15, 2335–2381.
41) Li, Y. & Camacho, P. (2004) Ca2+-dependent redox modulation of SERCA 2b by ERp57. J. Cell Biol., 164, 35–46.
42) Higo, T., Hattori, M., Nakamura, T., Natsume, T., Michikawa, T., & Mikoshiba, K. (2005) Subtype-specific and ER lumenal environment-dependent regulation of inositol 1,4,5-trisphosphate receptor Type 1 by ERp44. Cell, 120, 85–98.
43) Ushioda, R., Miyamoto, A., Inoue, M., Watanabe, S., Okumura, M., Maegawa, K.I., Uegaki, K., Fujii, S., Fukuda, Y., Umitsu, M., et al. (2016) Redox-assisted regulation of Ca2+ homeostasis in the endoplasmic reticulum by disulfide reductase ERdj5. Proc. Natl. Acad. Sci. USA, 113, E6055–E6063.
44) 潮田亮(2020)レドックス制御による小胞体恒常性維持機構の解明~還元反応の場としての小胞体~.生化学,92, 536–546.
45) Higa, A., Taouji, S., Lhomond, S., Jensen, D., Fernandez-Zapico, M.E., Simpson, J.C., Pasquet, J.M., Schekman, R., & Chevet, E. (2014) Endoplasmic reticulum stress-activated transcription factor ATF6 requires the disulfide isomerase PDIA5 to modulate chemoresistance. Mol. Cell. Biol., 34, 1839–1849.
46) Oka, O.B., Lith, M., Rudolf, J., Tungkum, W., Pringle, M.A., & Bulleid, N.J. (2019) ERp18 regulates activation of ATF6α during unfolded protein response. EMBO J., 38, 1–18.
47) Eletto, D., Eletto, D., Dersh, D., Gidalevitz, T., & Argon, Y. (2014) Protein disulfide isomerase A6 controls the decay of IRE1α signaling via disulfide-dependent association. Mol. Cell, 53, 562–576.
2024年11月26日 | カテゴリー:各種病因学, 癌の病態生理と治療学, 基礎知識/物理学、統計学、有機化学、数学、英語 |